La Forme du Sensible : Classification (partie 2)
Sciencylogie
AuteurRomain Piccollo
La classification scientifique, ou taxonomie, va bien au-delà d’une simple mise en ordre du vivant. Elle constitue un langage commun qui permet aux biologistes de nommer, comparer et comprendre la diversité des formes de vie. Des premiers catalogues antiques aux arbres phylogénétiques générés par algorithmes, cette organisation reflète une quête : celle de décrypter les liens de parenté et de retracer l’histoire évolutive des espèces.
Ce système révèle aussi les limites de la taxonomie classique, confrontée aux pièges des convergences évolutives et à la richesse souvent ignorée du microbiote. Face à ces défis, les chercheurs mobilisent aujourd’hui la génomique et l’intelligence artificielle, pour construire une vision plus fidèle et dynamique de la diversité biologique. Un domaine complexe que nous vous invitons à explorer.
Sommaire
- Qu’est-ce que la classification scientifique du vivant ?
- Quels critères utilise-t-on pour classer les êtres vivants ?
- En quoi la classification diffère-t-elle des outils d’identification ?
- Quels types de données les biologistes comparent-ils aujourd’hui ?
- Qu’est-ce qu’une espèce et comment l’identifie-t-on ?
- Que sont les espèces cryptiques et comment les repère-t-on ?
- Comment détecte-t-on des espèces sans même les observer ?
- Pourquoi la phylogénie est-elle devenue centrale dans la classification ?
- Pourquoi les rangs traditionnels posent-ils problème aujourd’hui ?
- Comment fonctionne la taxonomie intégrative ?
- Comment la technologie aide-t-elle à mieux classer le vivant ?
- Quels sont les enjeux éthiques et géopolitiques de la classification ?
- Quels sont les principaux défis de la taxonomie actuelle ?
- Pourquoi faut-il intégrer l’écologie et la fonction dans la classification ?
- Qu’est-ce que la phylodynamie et pourquoi est-elle importante ?
- À quoi ressemblera la classification du vivant dans le futur ?
Qu’est-ce que la classification scientifique du vivant ?
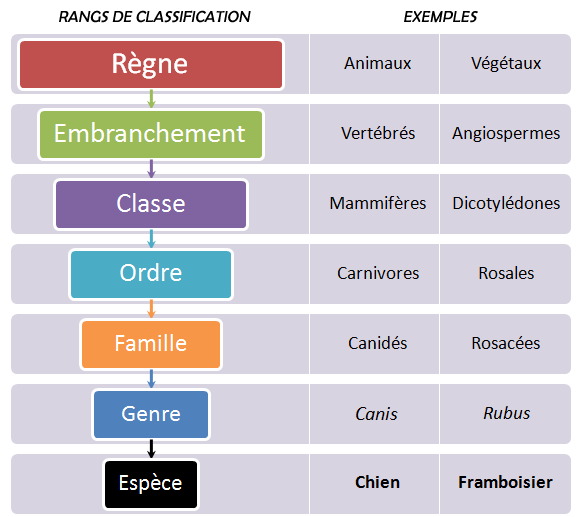
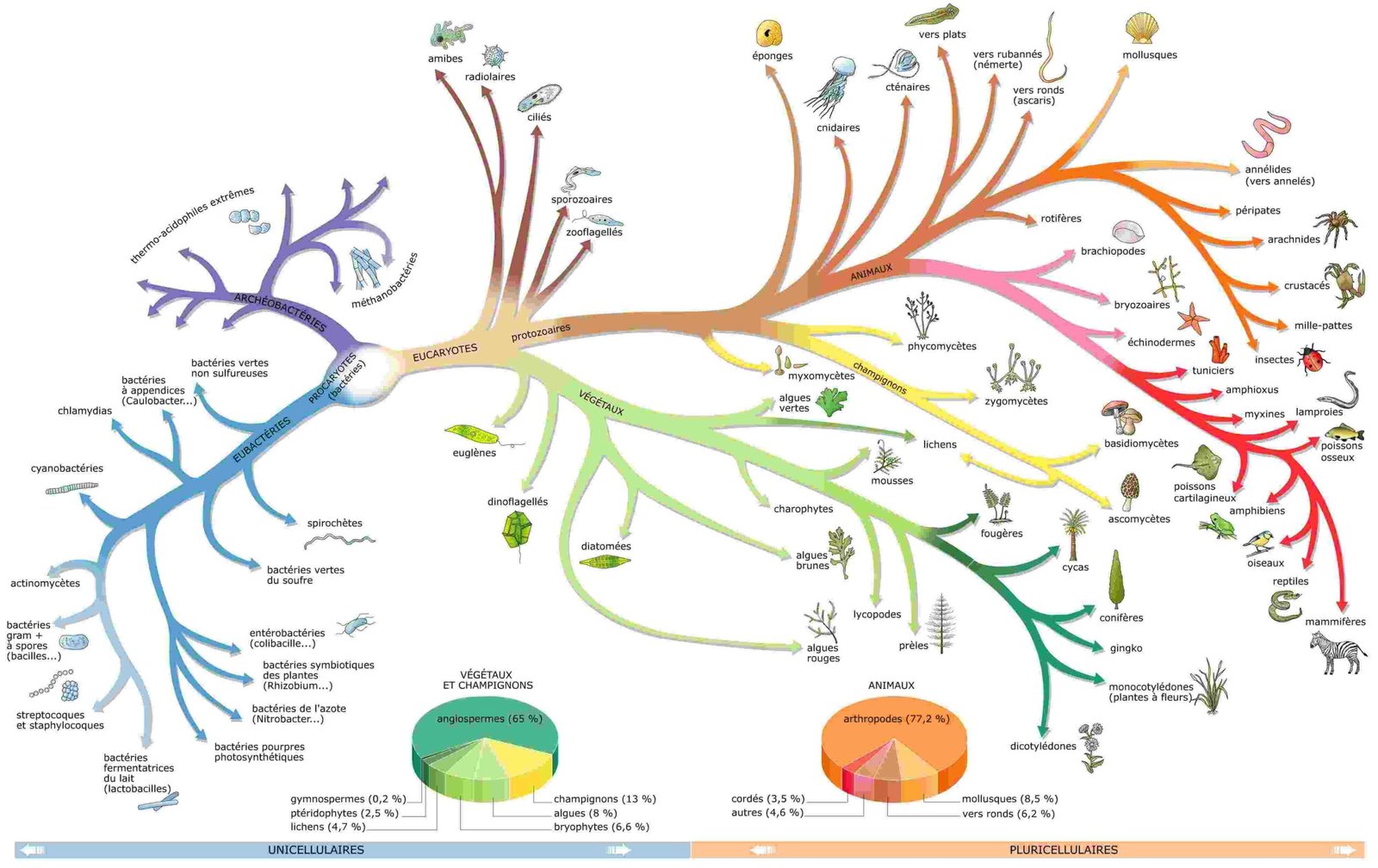
Classer les êtres vivants consiste à organiser la diversité du vivant en groupes distincts, selon des critères observables ou mesurables. La classification scientifique répartit ces organismes à différentes échelles (espèce, genre, famille) en fonction de caractères communs. Ces critères peuvent être morphologiques, génétiques ou écologiques.
Ce système facilite la nomination, la comparaison et l’identification des organismes grâce à une méthode partagée. Il établit aussi des liens entre les espèces, en intégrant leur histoire évolutive quand les données le permettent.
La taxonomie a dépassé une vision figée du vivant. Elle ne se limite plus à décrire, mais cherche à expliquer. Cette science s’appuie sur la phylogénie pour reconstituer les relations entre lignées et s’adapte aux progrès des connaissances, des outils et des découvertes.
À ce jour, les scientifiques ont décrit près de 1,9 million d’espèces. Parmi elles, seules 220 000 à 300 000 vivent en milieu marin, ce qui représente entre 12 et 15 % de la biodiversité connue. Ce catalogue comprend environ 1,6 million d’animaux, 350 000 espèces végétales et plusieurs dizaines de milliers de micro-organismes.
Le Catalogue de la Vie
Catalogue of Life est une base de données mondiale qui recense toutes les espèces vivantes connues, validées par des experts. Elle sert de référence taxonomique pour la recherche, la conservation et les politiques environnementales.
Taxonomie
Science qui classifie les êtres vivants en catégories hiérarchiques (espèce, genre, famille, etc.) selon leurs caractéristiques communes.
Quels critères utilise-t-on pour classer les êtres vivants ?
Au-delà de l’apparence, les scientifiques examinent plusieurs critères pour distinguer les espèces. La classification intègre la morphologie externe, l’anatomie interne, le comportement, le mode de reproduction, les caractéristiques écologiques ainsi que les données génétiques. Aujourd’hui, le séquençage de l’ADN tient une place centrale dans de nombreuses recherches.
Ces critères ne pèsent pas tous de la même façon, selon les groupes d’organismes ou le contexte d’étude. Les classifications actuelles reposent souvent sur un équilibre subtil entre différentes informations. Leur but : refléter au plus juste les liens évolutifs qui unissent les espèces.
En quoi la classification diffère-t-elle des outils d’identification ?
La taxonomie s’appuie sur des éléments visibles, mais aussi sur des critères plus abstraits. Les clés d’identification aident à reconnaître rapidement une espèce grâce à quelques caractéristiques simples. La classification, elle, vise à organiser le vivant dans son ensemble. Elle regroupe les organismes selon des liens de parenté ou des ressemblances structurelles. Cette démarche, plus théorique, se conçoit pour durer.
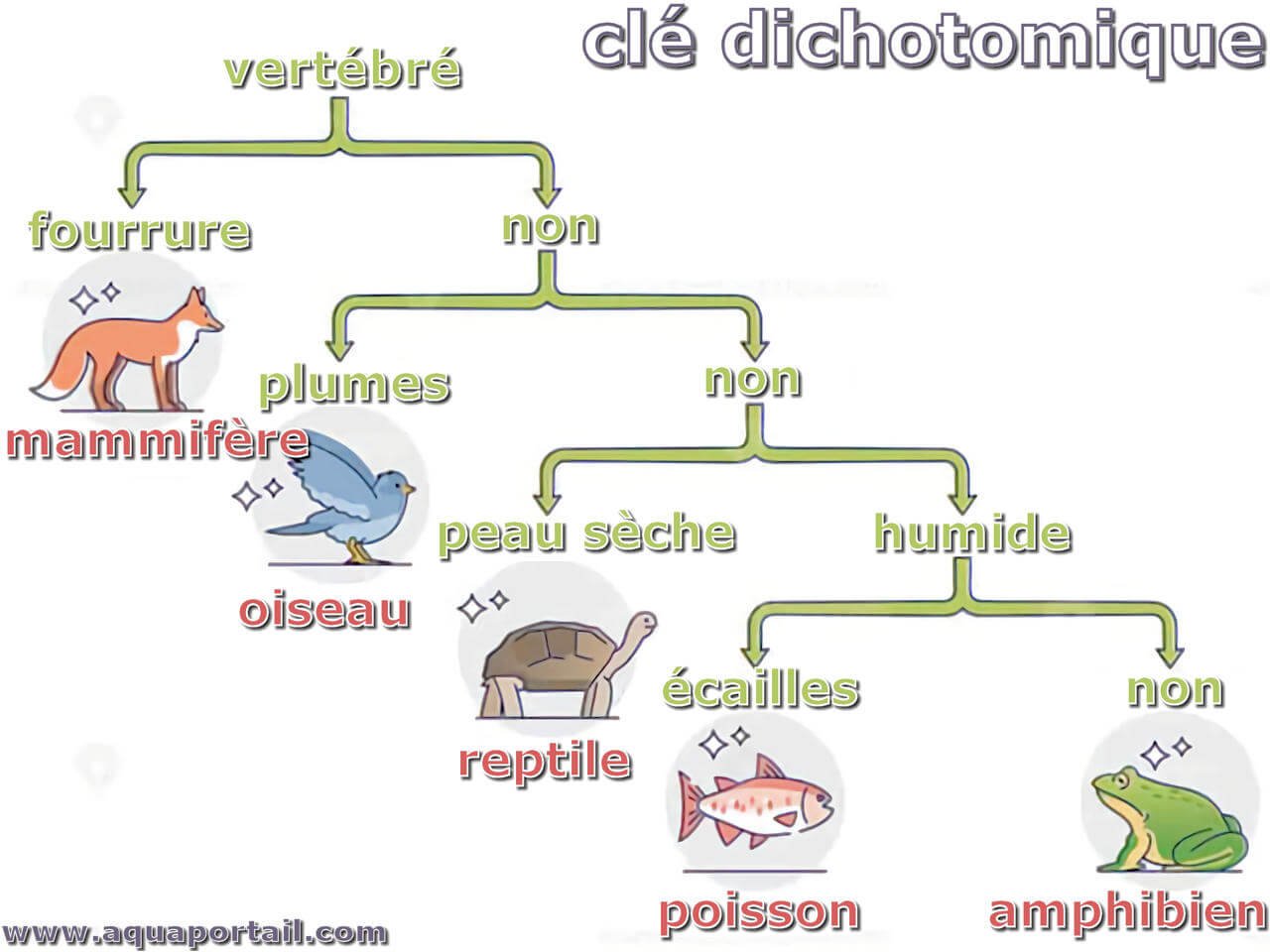
Parmi les outils les plus utilisés : les clés dichotomiques. Elles fonctionnent par choix successifs, opposant à chaque étape deux options, comme “avec ou sans poils”. Ce système guide pas à pas vers l’espèce recherchée. Ces clés restent largement employées, surtout en éducation et dans les programmes de sciences participatives.
Clé dichotomique
Série de couplets de questions liées les unes aux autres qui permet d’identifier un individu animal ou végétal selon son ordre, sa classe ou même son espèce.
Clé d’identification
Outil permettant d’identifier un taxon (espèce, genre, famille, etc.) en suivant une série de choix basés sur des caractéristiques observables.
Quels types de données les biologistes comparent-ils aujourd’hui ?
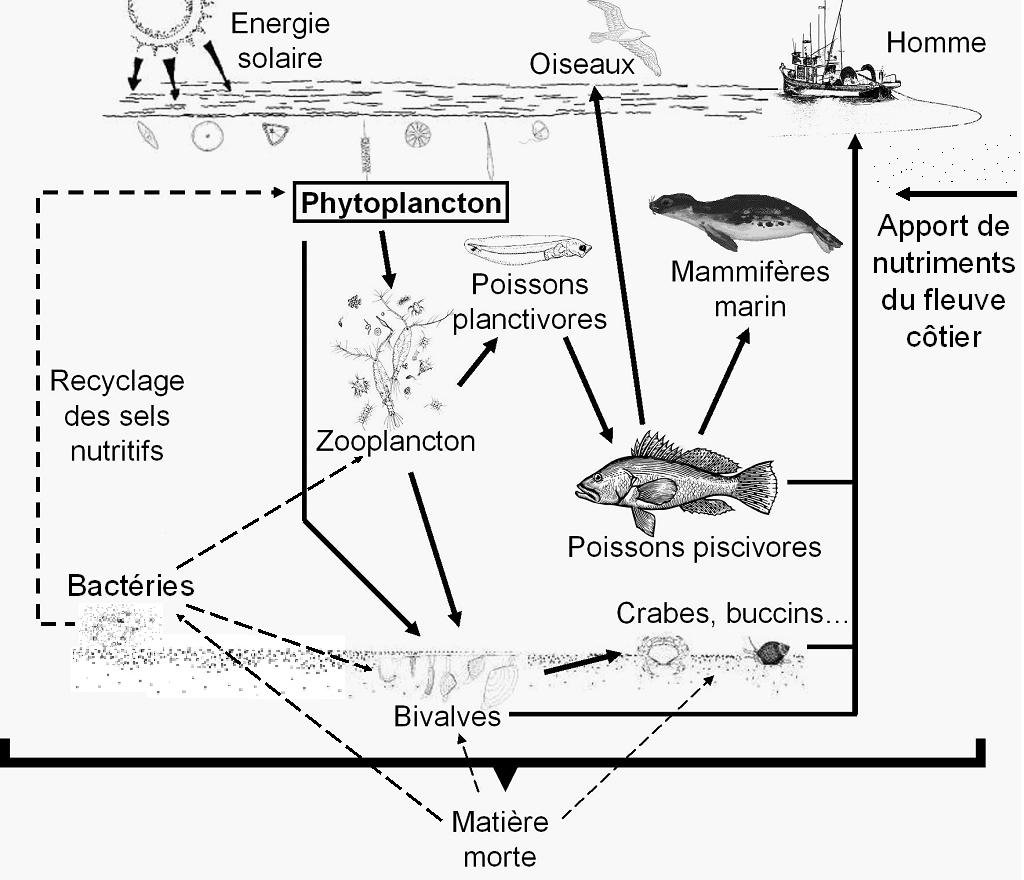
Pour saisir les liens entre les espèces, les biologistes dépassent désormais l’apparence. Ils croisent des données issues de plusieurs disciplines : séquences génétiques, formes anatomiques, comportements, habitats, et informations écologiques.
L’ADN révèle des ressemblances ou des différences invisibles à l’œil nu. Les comportements et les milieux de vie apportent un éclairage complémentaire sur l’histoire évolutive d’un organisme. Certaines espèces, éloignées morphologiquement, partagent pourtant des séquences génétiques proches. D’autres, proches au premier regard, appartiennent à des lignées nettement distinctes (exemple ci-dessous).
En combinant ces éléments, les chercheurs reconstruisent des arbres phylogénétiques plus précis. Cette approche globale éclaire l’évolution du vivant et la richesse de la biodiversité actuelle.
Qu’est-ce qu’une espèce et comment l’identifie-t-on ?
Le concept de la nomenclature binominale est relativement simple : chaque espèce vivante se voit attribuer un nom scientifique unique composé de deux éléments en latin : le nom du genre, suivi du nom de l’espèce. Par exemple, pour l’espèce humaine, le nom du genre est Homo et celui de l’espèce est sapiens.
Ce système permet de structurer plus efficacement nos connaissances sur les espèces et s’est avéré indispensable pour les premières représentations de l’arbre du vivant.
Espèce morphologique
Elle repose sur l’apparence. Des chercheurs regroupent les individus qui partagent des traits visibles, comme la taille, la forme ou la structure d’un organe. Cette définition s’utilise surtout pour les fossiles ou les organismes peu connus, quand les données génétiques manquent.
Espèce écologique
Elle regroupe les organismes qui occupent une même niche écologique. Ils vivent dans un même type d’environnement et utilisent les mêmes ressources. Cette approche prend en compte les fonctions des espèces dans un écosystème.
Espèce phylogénétique
Elle correspond à la plus petite lignée identifiable qui possède une combinaison unique de caractères. Ces caractères sont souvent génétiques. Ce concept gagne du terrain avec les progrès en génomique, car il permet de détecter des distinctions invisibles à l’œil nu.
En pratique, la délimitation des espèces dépend des méthodes disponibles et des données accessibles. Les frontières entre espèces ne sont pas toujours nettes, ce qui peut entraîner des désaccords entre chercheurs. C’est pourquoi il existe une multitude de concepts d’espèces, chacun ayant ses avantages et ses limites.
Que sont les espèces cryptiques et comment les repère-t-on ?
Les espèces cryptiques rassemblent des organismes génétiquement distincts, souvent incapables de se reproduire entre eux. Leur morphologie, très proche, rend leur distinction difficile à l’œil nu. Cette similitude peut les faire passer pour une seule espèce, sous-estimant ainsi la richesse réelle du vivant.
Ce système permet de structurer plus efficacement nos connaissances sur les espèces et s’est avéré indispensable pour les premières représentations de l’arbre du vivant.
Les analyses génétiques
Le séquençage de l’ADN, en particulier grâce au barcoding moléculaire (codage à barres de l’ADN), révèle des différences génétiques marquées entre individus qui paraissent identiques à l’œil nu.
Études comportementales et écologiques
Des différences dans les chants, les rythmes de vie ou les préférences écologiques permettent parfois de distinguer des espèces proches, invisibles autrement.
Bioacoustique
L’analyse des sons émis, comme les stridulations chez certaines fourmis, aide à discerner des espèces cryptiques invisibles autrement.
Morphométrie fine
Des mesures précises de structures anatomiques, souvent imperceptibles à l’œil nu, mettent au jour des différences subtiles entre espèces.
Des exemples notables
- Taupe aquitaine (Talpa aquitania) : longtemps confondue avec la taupe européenne, cette espèce a été identifiée grâce à des analyses génétiques et morphologiques détaillées.
- Murin cryptique (Myotis crypticus) : cette chauve-souris, morphologiquement proche du Murin de Natterer, a été reconnue comme une espèce distincte en 2019 grâce à des études génétiques.
- Hyalella azteca : ce crustacé d’eau douce, utilisé comme bioindicateur, s’est révélé être un complexe de plus de 30 espèces cryptiques identifiées par des analyses génétiques.
La découverte d’espèces cryptiques souligne l’importance des méthodes modernes en taxonomie pour saisir avec précision la biodiversité. Elle révèle la nécessité d’une approche intégrative, mêlant génétique, écologie et morphologie, afin de dévoiler la richesse cachée du vivant.
Il faut aussi distinguer clairement les espèces cryptiques des cryptides. Ces derniers sont des créatures dont l’existence repose sur des témoignages ou des légendes, sans confirmation scientifique. Le monstre du Loch Ness ou le yéti en sont des exemples célèbres. Fascinants, ces cryptides appartiennent plutôt au folklore et à la cryptozoologie qu’à la taxonomie scientifique.
Barcoding moléculaire
Une méthode qui permet d’identifier une espèce vivante en analysant un tout petit fragment de son ADN, comme on le ferait avec un code-barres en magasin. Ce “code génétique” unique permet de reconnaître rapidement une espèce, même à partir d’un simple poil, d’une plume ou d’un reste de feuille.
Stridulations
Des sons produits par certains insectes, comme les criquets, les grillons ou les sauterelles, en frottant des parties de leur corps entre elles. Ces sons servent souvent à attirer un partenaire, marquer leur territoire ou se défendre.
Comment détecte-t-on des espèces sans même les observer ?
L’ADN environnemental, ou eDNA, révèle la présence d’organismes grâce aux fragments d’ADN qu’ils déposent dans leur milieu. Cette méthode s’avère particulièrement utile dans l’eau ou dans des milieux difficiles d’accès.
Elle permet d’établir des inventaires d’espèces, de suivre la biodiversité et de repérer des espèces rares ou envahissantes. Néanmoins, elle ne remplace pas les observations classiques indispensables à la description formelle des espèces.
Classer ce qui échappe à l’œil exige un savoir-faire spécifique. Chez les micro-organismes, la reproduction sans sexuation, le transfert horizontal de gènes, ou l’impossibilité de les cultiver brouillent les pistes. Pour s’en sortir, les chercheurs misent sur la phylogénie et la génomique.
ADN environnemental (ADNe)
Fragments d’ADN laissés par les organismes dans leur environnement (eau, sol, air), permettant de détecter leur présence sans les observer directement.
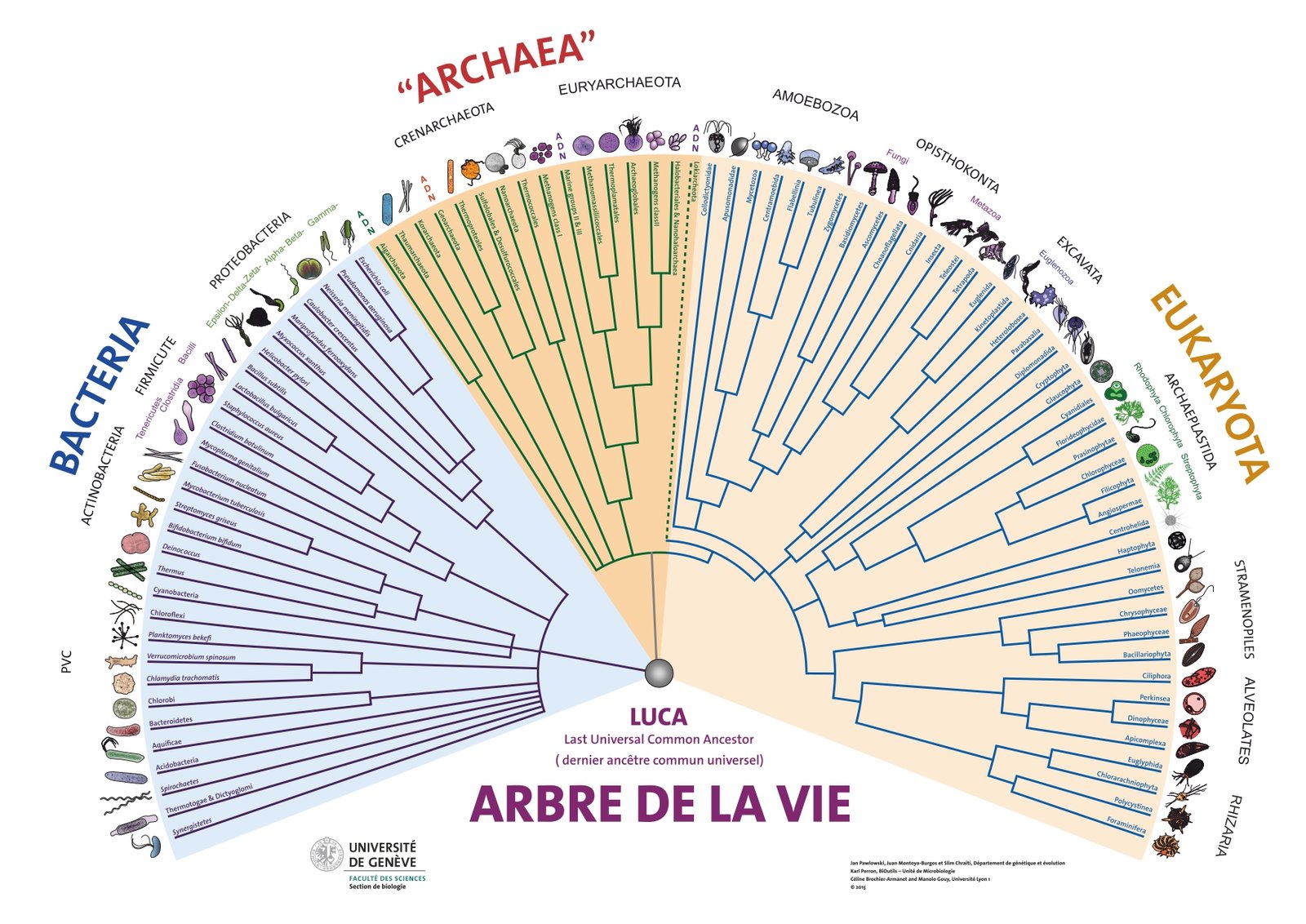
Pourquoi la phylogénie est-elle devenue centrale dans la classification ?
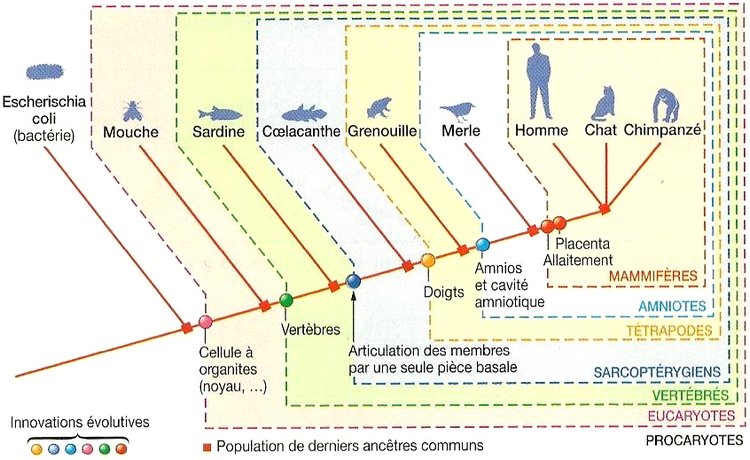
La phylogénie trace les relations évolutives entre espèces sous la forme d’un arbre, où chaque nœud représente un ancêtre commun. En systématique moderne, l’objectif est de regrouper des espèces au sein de clades.
Cette méthode cherche à rendre justice aux parcours évolutifs. Elle conduit parfois à modifier ou rejeter des classifications anciennes, établies seulement en fonction de formes analogues ou de pratiques historiques, si celles-ci ne correspondent pas aux liens véritables détectés.
Clade
Un groupe d’espèces qui descend toutes d’un même ancêtre commun, et qui inclut cet ancêtre ainsi que l’ensemble de ses descendants.
Phylogénie
C’est l’étude des relations évolutives entre les espèces ou les groupes d’organismes. Elle cherche à reconstruire leur histoire commune en établissant des arbres généalogiques appelés arbres phylogénétiques, qui montrent comment les espèces sont liées par la descendance à partir d’ancêtres communs.
Groupe monophylétique
Ensemble d’espèces comprenant un ancêtre commun et tous ses descendants.
Pourquoi les rangs traditionnels posent-ils problème aujourd’hui ?
Le classement par règnes, classes, ordres, hérité de l’histoire des sciences, ne correspond pas toujours à la vraie diversité et aux parcours évolutifs. Certains spécialistes recommandent d’employer de ces fameux clades, qui forment des ensembles définis uniquement à partir d’un ancêtre commun, sans les placer dans une hiérarchie rigide. Cette approche apparaît plus fidèle au vivant.
Le débat persiste. Les classes et ordres traditionnels restent largement utilisés, car ils facilitent la lecture des données et assurent la cohérence avec les bases existantes. Autrement dit, les deux systèmes cohabitent : les clades apportent une fidélité phylogénétique, tandis que les rangs permettent lisibilité et compatibilité.
Comment fonctionne la taxonomie intégrative ?
Cette discipline moderne vise à croiser divers types de données (forme, ADN, comportements, habitat) pour définir les espèces avec davantage de précision. Autrefois, identifier une espèce reposait souvent sur un seul critère, généralement morphologique. Aujourd’hui, on combine ce dernier à d’autres sources d’information, ce qui réduit les risques d’erreur.
Prenons un cas concret : deux organismes peuvent paraître identiques à l’œil, mais révéler des divergences génétiques évidentes. Ces écarts suggèrent qu’il s’agit bien de deux espèces distinctes. C’est grâce à une alliance de critères que la taxonomie devient plus fiable, plus riche et plus pertinente.
Comment la technologie aide-t-elle à mieux classer le vivant ?
L’arrivée des outils de capture 3D (microCT, photogrammétrie) permet d’explorer la structure interne et externe des organismes sans les altérer. Des dizaines de milliers de points de données morphologiques sont analysés statistiquement, enrichissant les comparaisons entre espèces.
À cela s’ajoute l’intelligence artificielle : les réseaux neuronaux détectent automatiquement des motifs et classent les images, qu’il s’agisse d’insectes, de champignons ou de plantes. Ce duo 3D‑IA multiplie la précision des identifications et multiplie la cadence de traitement au-delà de ce qu’il était possible manuellement.
Fruit de ces innovations, la taxonomie gagne en rigueur (précision des mesures, validité des diagnostics) et en dynamisme, avec la capacité à intégrer en continu les nouvelles données.
MicroCT
Une technique d’imagerie 3D qui permet de visualiser en détail l’intérieur d’un organisme sans l’abîmer. Comme un scanner médical, mais en plus précis, il révèle la structure fine des os, tissus ou coquilles, pour aider à comparer les espèces de façon très précise.
Photogrammétrie
Une technique qui utilise des photos prises sous différents angles pour créer un modèle 3D précis d’un objet ou d’un lieu. Elle permet de mesurer et de reconstruire la forme et les détails sans toucher ni abîmer ce qu’on photographie.
Quels sont les enjeux éthiques et géopolitiques de la classification ?
L’accès aux ressources génétiques s’organise autour d’accords internationaux, à l’image du Protocole de Nagoya. Celui-ci impose un consentement préalable et l’établissement de modalités équitables de partage, y compris dans le champ de la recherche taxonomique. Ce régime vise à garantir que ceux qui fournissent les échantillons reçoivent des bénéfices justes.
Pourtant, certains dénoncent un déséquilibre persistant : les pays riches sur le plan technologique prennent le pas sur les nations riches en biodiversité. Les débats autour de l’usage des données génomiques numériques (DSI), notamment à la COP16, illustrent cet affrontement entre acteurs du Nord et du Sud.
Dans ce contexte, les ressources en ligne constituent une avancée décisive. Des plateformes telles que ITIS, World Flora Online, ZooBank, FishBase ou iNaturalist libèrent l’accès à des bases taxonomiques, à des images, à des données de répartition. Elles servent la recherche, l’éducation et la conservation, tout en contribuant à corriger les inégalités d’accès à l’information scientifique à l’échelle mondiale.
Protocole de Nagoya
Accord international visant à assurer le partage juste et équitable des avantages découlant de l’utilisation des ressources génétiques.
Quels sont les principaux défis de la taxonomie actuelle ?
La recherche taxonomique traverse une crise silencieuse : dans de nombreux groupes d’organismes, il manque des spécialistes. Ce déficit, connu sous le nom d’impédiment taxonomique, freine l’identification des espèces, notamment dans les régions les plus riches en biodiversité. Faute de relais, certaines connaissances s’effacent, d’autres stagnent dans des tiroirs.
À cette pénurie s’ajoute une autre difficulté : l’explosion des données. Séquences génétiques, images à haute résolution, relevés d’observations… les informations s’accumulent à une vitesse vertigineuse. Les classer, les comparer, les rendre accessibles exige des outils informatiques robustes et une standardisation rigoureuse. Ces solutions émergent peu à peu, mais leur mise en œuvre reste complexe et morcelée.
Pourquoi faut-il intégrer l’écologie et la fonction dans la classification ?
Classer selon les seuls liens évolutifs ne rend pas justice à toute la richesse du vivant. Cette démarche peut passer sous silence le rôle d’une espèce dans son écosystème ou ses spécificités physiologiques. Ajouter ces données apporte une vision plus vaste du monde vivant. L’histoire évolutive ne décrit pas tout. Pour saisir une espèce, il faut examiner ses fonctions, sa place et ses interactions avec le milieu.
Cette méthode met en lumière les relations entre espèces, anticipe les conséquences des perturbations écologiques et oriente les décisions de préservation. Répartir les organismes dans une classification fonctionnelle et écologique aide à analyser réseaux biologiques, flux de matière et d’énergie ainsi que la capacité des habitats à absorber les chocs. Elle demeure indispensable en biogéographie, discipline qui scrute la répartition géographique de la biodiversité.
Clade
Groupe d’organismes comprenant un ancêtre commun et tous ses descendants, équivalent à un groupe monophylétique.
Qu’est-ce que la phylodynamie et pourquoi est-elle importante ?
La phylodynamie est une discipline qui croise trois domaines : la génétique, l’épidémiologie et l’évolution. Elle cherche à comprendre comment les virus ou les bactéries changent, se transmettent et circulent dans les populations.
En étudiant leur évolution génétique au fil du temps, elle permet de reconstituer les chaînes de transmission, de mesurer la vitesse de contagion et de localiser les foyers d’infection. Pendant la crise du COVID-19, la phylodynamie a joué une importance cruciale : elle a aidé à retracer l’histoire du virus, à détecter les variants et à orienter les décisions de santé publique.
Mais cette approche ne se limite pas aux maladies humaines. Elle sert aussi à étudier les réponses rapides d’animaux, de plantes ou de microbes face à des pressions comme le changement climatique, les antibiotiques ou la destruction des habitats. Une manière, en somme, de suivre la vie en mouvement.
Nextstrain
Nextstrain est un outil phare de la phylodynamie appliquée au SARS-CoV-2 : il permet de visualiser l’évolution du virus, de suivre sa transmission et de guider la réponse sanitaire à travers des analyses phylogénétiques en temps réel.
À quoi ressemblera la classification du vivant dans le futur ?
À ce jour, seuls 20 % des espèces vivant sur Terre ont été décrites. Le reste échappe encore à notre regard. Mais les choses bougent. Grâce aux avancées du séquençage génétique, à l’intelligence artificielle et au partage massif de données, la manière de classer les espèces évolue. Fini les systèmes figés : place à une classification vivante, capable d’intégrer les découvertes au fil de leur apparition.
Pour autant, les méthodes traditionnelles ne disparaissent pas. Les nomenclatures rigoureuses, les descriptions morphologiques et les grandes collections naturalistes conservent une importance certaine dans l’étude de la biodiversité.
C’est dans cet esprit qu’est né le métaprogramme DIGIT-BIO, piloté par le CNRS. Son objectif : faire dialoguer biologie, numérique et modélisation pour repenser la classification. En croisant des données massives et des outils de pointe, ce projet aide à identifier des espèces encore inconnues et à redessiner l’arbre du vivant.
Une nouvelle taxonomie se dessine ainsi : plus fluide, plus réactive, et surtout, plus fidèle à la complexité du monde vivant.